ga
Trova il minimo della funzione usando l'algoritmo genetico
Sintassi
Descrizione
x = ga(fun,nvars)x, per la funzione obiettivo, fun . nvars è la dimensione (numero di variabili di progettazione) di fun .
Nota
Passing Extra Parameters spiega come passare parametri aggiuntivi alla funzione obiettivo e alle funzioni di vincolo non lineare, se necessario.
x = ga(fun,nvars,A,b,Aeq,beq,lb,ub,nonlcon)nonlcon . La funzione nonlcon accetta x e restituisce i vettori C e Ceq, che rappresentano rispettivamente le disuguaglianze e le uguaglianze non lineari. ga minimizza fun in modo che C(x) ≤ 0 e Ceq(x) = 0 . (Impostare lb=[] e ub=[] se non esistono limiti.)
x = ga(fun,nvars,A,b,Aeq,beq,lb,ub,nonlcon,intcon)x = ga(fun,nvars,A,b,Aeq,beq,lb,ub,nonlcon,intcon,options)intcon assumano valori interi.
Nota
In presenza di vincoli interi, ga non accetta vincoli di uguaglianza non lineare, ma solo vincoli di disuguaglianza non lineare.
Esempi
Quando si esegue questo esempio, viene incluso il file ps_example.m. Rappresenta graficamente la funzione.
xi = linspace(-6,2,300); yi = linspace(-4,4,300); [X,Y] = meshgrid(xi,yi); Z = ps_example([X(:),Y(:)]); Z = reshape(Z,size(X)); surf(X,Y,Z,'MeshStyle','none') colormap 'jet' view(-26,43) xlabel('x(1)') ylabel('x(2)') title('ps\_example(x)')
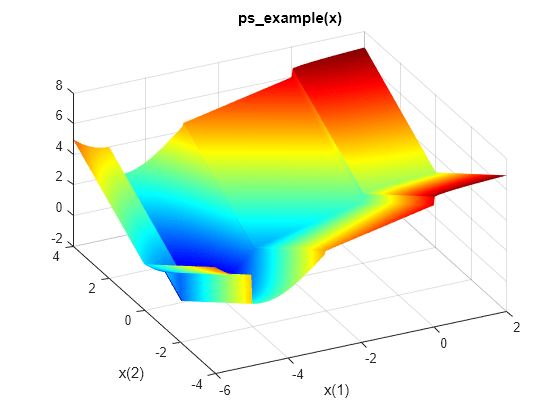
Trova il minimo di questa funzione usando ga .
rng default % For reproducibility x = ga(@ps_example,2)
ga stopped because the average change in the fitness value is less than options.FunctionTolerance.
x = 1×2
-4.6793 -0.0860
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example sulla regione x(1) + x(2) >= 1 e x(2) <= 5 + x(1). Questa funzione è inclusa quando si esegue questo esempio.
Per prima cosa, convertiamo i due vincoli di disuguaglianza nella forma matriciale A*x <= b . In altre parole, prendi le variabili x sul lato sinistro della disuguaglianza e rendi entrambe le disuguaglianze minori o uguali:
-x(1) -x(2) <= -1
-x(1) + x(2) <= 5
A = [-1,-1;
-1,1];
b = [-1;5];Risolvi il problema vincolato usando ga .
rng default % For reproducibility fun = @ps_example; x = ga(fun,2,A,b)
ga stopped because the average change in the fitness value is less than options.FunctionTolerance.
x = 1×2
0.9990 0.0000
I vincoli sono soddisfatti entro il valore predefinito della tolleranza del vincolo, 1e-3. Per vederlo, calcola A*x' - b, che dovrebbe avere componenti negative.
disp(A*x' - b)
0.0010 -5.9990
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example sulla regione x(1) + x(2) >= 1 e x(2) == 5 + x(1). Questa funzione è inclusa quando si esegue questo esempio.
Per prima cosa, convertiamo i due vincoli nella forma matriciale A*x <= b e Aeq*x = beq. In altre parole, prendi le variabili x sul lato sinistro delle espressioni e trasforma la disuguaglianza nella forma minore o uguale:
-x(1) -x(2) <= -1
-x(1) + x(2) == 5
A = [-1 -1]; b = -1; Aeq = [-1 1]; beq = 5;
Risolvi il problema vincolato usando ga .
rng default % For reproducibility fun = @ps_example; x = ga(fun,2,A,b,Aeq,beq)
ga stopped because the average change in the fitness value is less than options.FunctionTolerance.
x = 1×2
-2.0005 2.9995
Verificare che i vincoli siano soddisfatti entro il valore predefinito di ConstraintTolerance, 1e-3.
disp(A*x' - b)
1.0000e-03
disp(Aeq*x' - beq)
8.5897e-09
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example sulla regione x(1) + x(2) >= 1 e x(2) == 5 + x(1). Quando si esegue questo esempio, viene inclusa la funzione ps_example. Inoltre, impostare i limiti 1 <= x(1) <= 6 e -3 <= x(2) <= 8.
Per prima cosa, convertiamo i due vincoli lineari nella forma matriciale A*x <= b e Aeq*x = beq. In altre parole, prendi le variabili x sul lato sinistro delle espressioni e trasforma la disuguaglianza nella forma minore o uguale:
-x(1) -x(2) <= -1
-x(1) + x(2) == 5
A = [-1 -1]; b = -1; Aeq = [-1 1]; beq = 5;
Imposta i limiti lb e ub .
lb = [1 -3]; ub = [6 8];
Risolvi il problema vincolato usando ga .
rng default % For reproducibility fun = @ps_example; x = ga(fun,2,A,b,Aeq,beq,lb,ub)
ga stopped because the average change in the fitness value is less than options.FunctionTolerance.
x = 1×2
1.0000 6.0000
Verificare che i vincoli lineari siano soddisfatti entro il valore predefinito di ConstraintTolerance, 1e-3.
disp(A*x' - b)
-6.0000
disp(Aeq*x' - beq)
-7.9765e-08
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example sulla regione e . Quando si esegue questo esempio, viene inclusa la funzione ps_example.
Per farlo, utilizzare la funzione ellipsecons.m che restituisce il vincolo di disuguaglianza nel primo output, c, e il vincolo di uguaglianza nel secondo output, ceq. Quando si esegue questo esempio, viene inclusa la funzione ellipsecons. Esaminare il codice ellipsecons.
type ellipseconsfunction [c,ceq] = ellipsecons(x) c = 2*x(1)^2 + x(2)^2 - 3; ceq = (x(1)+1)^2 - (x(2)/2)^4;
Includere una funzione handle per ellipsecons come argomento nonlcon.
nonlcon = @ellipsecons; fun = @ps_example; rng default % For reproducibility x = ga(fun,2,[],[],[],[],[],[],nonlcon)
Optimization finished: average change in the fitness value less than options.FunctionTolerance and constraint violation is less than options.ConstraintTolerance.
x = 1×2
-0.9766 0.0362
Verificare che i vincoli non lineari siano soddisfatti in x . I vincoli sono soddisfatti quando c ≤ 0 e ceq = 0 entro il valore predefinito di ConstraintTolerance, 1e-3 .
[c,ceq] = nonlcon(x)
c = -1.0911
ceq = 5.4645e-04
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example sulle regioni x(1) + x(2) >= 1 e x(2) == 5 + x(1) utilizzando una tolleranza di vincolo inferiore a quella predefinita. Quando si esegue questo esempio, viene inclusa la funzione ps_example.
Per prima cosa, convertiamo i due vincoli nella forma matriciale A*x <= b e Aeq*x = beq. In altre parole, prendi le variabili x sul lato sinistro delle espressioni e trasforma la disuguaglianza nella forma minore o uguale:
-x(1) -x(2) <= -1
-x(1) + x(2) == 5
A = [-1 -1]; b = -1; Aeq = [-1 1]; beq = 5;
Per ottenere una soluzione più accurata, impostare una tolleranza di vincolo pari a 1e-6. Per monitorare i progressi del risolutore, imposta una funzione di grafico.
options = optimoptions('ga','ConstraintTolerance',1e-6,'PlotFcn', @gaplotbestf);
Risolvere il problema di minimizzazione.
rng default % For reproducibility fun = @ps_example; x = ga(fun,2,A,b,Aeq,beq,[],[],[],options)
ga stopped because the average change in the fitness value is less than options.FunctionTolerance.

x = 1×2
-2.0000 3.0000
Verificare che i vincoli lineari siano soddisfatti entro 1e-6 .
disp(A*x' - b)
9.9809e-07
disp(Aeq*x' - beq)
-7.3589e-08
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example soggetta al vincolo che x(1) sia un numero intero. Questa funzione è inclusa quando si esegue questo esempio.
intcon = 1; rng default % For reproducibility fun = @ps_example; A = []; b = []; Aeq = []; beq = []; lb = []; ub = []; nonlcon = []; x = ga(fun,2,A,b,Aeq,beq,lb,ub,nonlcon,intcon)
ga stopped because the average change in the penalty function value is less than options.FunctionTolerance and the constraint violation is less than options.ConstraintTolerance.
x = 1×2
-5.0000 -0.0834
Utilizzare l'algoritmo genetico per minimizzare un problema non lineare vincolato a numeri interi. Ottenere sia la posizione del minimo che il valore minimo della funzione. Quando si esegue questo esempio, viene inclusa la funzione obiettivo ps_example.
intcon = 1; rng default % For reproducibility fun = @ps_example; A = []; b = []; Aeq = []; beq = []; lb = []; ub = []; nonlcon = []; [x,fval] = ga(fun,2,A,b,Aeq,beq,lb,ub,nonlcon,intcon)
ga stopped because the average change in the penalty function value is less than options.FunctionTolerance and the constraint violation is less than options.ConstraintTolerance.
x = 1×2
-5.0000 -0.0834
fval = -1.8344
Confronta questo risultato con la soluzione del problema senza vincoli.
[x,fval] = ga(fun,2)
ga stopped because the average change in the fitness value is less than options.FunctionTolerance.
x = 1×2
-4.6906 -0.0078
fval = -1.9918
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example vincolata ad avere valori interi x(1). Quando si esegue questo esempio, viene inclusa la funzione ps_example. Per comprendere il motivo per cui il risolutore si è fermato e come ga ha cercato un minimo, ottenere i risultati exitflag e output. Inoltre, tracciare il grafico del valore minimo osservato della funzione obiettivo man mano che il risolutore procede.
intcon = 1; rng default % For reproducibility fun = @ps_example; A = []; b = []; Aeq = []; beq = []; lb = []; ub = []; nonlcon = []; options = optimoptions('ga','PlotFcn', @gaplotbestf); [x,fval,exitflag,output] = ga(fun,2,A,b,Aeq,beq,lb,ub,nonlcon,intcon,options)

ga stopped because the average change in the penalty function value is less than options.FunctionTolerance and the constraint violation is less than options.ConstraintTolerance.
x = 1×2
-5.0000 -0.0834
fval = -1.8344
exitflag = 1
output = struct with fields:
problemtype: 'integerconstraints'
rngstate: [1x1 struct]
generations: 86
funccount: 3311
message: 'ga stopped because the average change in the penalty function value is less than options.FunctionTolerance and ...'
maxconstraint: 0
hybridflag: []
Utilizzare l'algoritmo genetico per minimizzare la funzione ps_example vincolata ad avere valori interi x(1). Quando si esegue questo esempio, viene inclusa la funzione ps_example. Ottenere tutti gli output, inclusa la popolazione finale e il vettore dei punteggi.
intcon = 1; rng default % For reproducibility fun = @ps_example; A = []; b = []; Aeq = []; beq = []; lb = []; ub = []; nonlcon = []; [x,fval,exitflag,output,population,scores] = ga(fun,2,A,b,Aeq,beq,lb,ub,nonlcon,intcon);
ga stopped because the average change in the penalty function value is less than options.FunctionTolerance and the constraint violation is less than options.ConstraintTolerance.
Esaminare i primi 10 membri della popolazione finale e i punteggi corrispondenti. Si noti che x(1) ha valore intero per tutti i membri della popolazione. L'algoritmo intero ga genera solo popolazioni fattibili a livello intero.
disp(population(1:10,:))
1.0e+03 *
-0.0050 -0.0001
-0.0050 -0.0001
-1.6420 0.0027
-1.5070 0.0010
-0.4540 0.0104
-0.2530 -0.0011
-0.1210 -0.0003
-0.1040 0.1314
-0.0140 -0.0010
0.0160 -0.0002
disp(scores(1:10))
1.0e+06 *
-0.0000
-0.0000
2.6798
2.2560
0.2016
0.0615
0.0135
0.0099
0.0001
0.0000
Argomenti di input
Funzione obiettivo, specificata come funzione handle o nome di funzione. Scrivere la funzione obiettivo in modo che accetti un vettore riga di lunghezza nvars e restituisca un valore scalare.
Quando l'opzione 'UseVectorized' è true, scrivere fun per accettare una matrice pop per nvars, dove pop è la dimensione attuale della popolazione. In questo caso, fun restituisce un vettore della stessa lunghezza di pop contenente i valori della funzione di fitness. Assicurarsi che fun non assuma alcuna dimensione particolare per pop, poiché ga può superare un singolo membro di una popolazione anche in un calcolo vettorializzato.
Esempio fun = @(x)(x-[4,2]).^2
Tipi di dati: char | function_handle | string
Numero di variabili, specificato come numero intero positivo. Il risolutore passa i vettori riga di lunghezza nvars a fun.
Esempio 4
Tipi di dati: double
Vincoli di disuguaglianza lineare, specificati come matrice reale. A è una matrice M per nvars, dove M è il numero di disuguaglianze.
A codifica le disuguaglianze lineari M
A*x <= b,
dove x è il vettore colonna delle variabili nvars x(:) e b è un vettore colonna con elementi M.
Ad esempio, per specificare
x1 + 2 x2 ≤ 10
3 x1 + 4 x2 ≤ 20
5 x1 + 6 x2 ≤ 30,
dare questi vincoli:
A = [1,2;3,4;5,6]; b = [10;20;30];
Esempio Per specificare che la somma delle variabili di controllo sia pari o inferiore a 1, specificare i vincoli A = ones(1,N) e b = 1.
Tipi di dati: double
Vincoli di disuguaglianza lineare, specificati come vettore reale. b è un vettore di elementi M correlato alla matrice A. Se si passa b come vettore riga, i risolutori convertono internamente b nel vettore colonna b(:).
b codifica le disuguaglianze lineari M
A*x <= b,
dove x è il vettore colonna delle variabili N x(:) e A è una matrice di dimensione M per N .
Ad esempio, per specificare
x1 + 2 x2 ≤ 10
3 x1 + 4 x2 ≤ 20
5 x1 + 6 x2 ≤ 30,
dare questi vincoli:
A = [1,2;3,4;5,6]; b = [10;20;30];
Esempio Per specificare che la somma delle variabili di controllo sia pari o inferiore a 1, specificare i vincoli A = ones(1,N) e b = 1.
Tipi di dati: double
Vincoli di uguaglianza lineare, specificati come matrice reale. Aeq è una matrice Me per nvars, dove Me è il numero di uguaglianze.
Aeq codifica le uguaglianze lineari Me
Aeq*x = beq,
dove x è il vettore colonna delle variabili N x(:) e beq è un vettore colonna con elementi Me.
Ad esempio, per specificare
x1 + 2 x2 + 3 x3 = 10
2 x1 + 4 x2 + x3 = 20,
dare questi vincoli:
Aeq = [1,2,3;2,4,1]; beq = [10;20];
Esempio Per specificare che la somma delle variabili di controllo sia 1, specificare i vincoli Aeq = ones(1,N) e beq = 1.
Tipi di dati: double
Vincoli di uguaglianza lineare, specificati come vettore reale. beq è un vettore di elementi Me correlato alla matrice Aeq. Se si passa beq come vettore riga, i risolutori convertono internamente beq nel vettore colonna beq(:).
beq codifica le uguaglianze lineari Me
Aeq*x = beq,
dove x è il vettore colonna delle variabili N x(:) e Aeq è una matrice di dimensione Meq per N .
Ad esempio, per specificare
x1 + 2 x2 + 3 x3 = 10
2 x1 + 4 x2 + x3 = 20,
dare questi vincoli:
Aeq = [1,2,3;2,4,1]; beq = [10;20];
Esempio Per specificare che la somma delle variabili di controllo sia 1, specificare i vincoli Aeq = ones(1,N) e beq = 1.
Tipi di dati: double
Limiti inferiori, specificati come array reale o array di double. lb rappresenta i limiti inferiori elemento per elemento in lb ≤ x ≤ ub .
Internamente, ga converte un array lb nell'array lb(:).
Esempio lb = [0;-Inf;4] significa x(1) ≥ 0, x(3) ≥ 4 .
Tipi di dati: double
Limiti superiori, specificati come array reale o array di double. ub rappresenta i limiti superiori elemento per elemento in lb ≤ x ≤ ub.
Internamente, ga converte un array ub nell'array ub(:).
Esempio ub = [Inf;4;10] significa x(2) ≤ 4, x(3) ≤ 10 .
Tipi di dati: double
Vincoli non lineari, specificati come funzione handle o nome di funzione. nonlcon è una funzione che accetta un vettore o un array x e restituisce due array, c(x) e ceq(x).
c(x)è la matrice dei vincoli di disuguaglianza non lineare inx.gatenta di soddisfarec(x) <= 0per tutte le voci di
c.ceq(x)è l'array di vincoli di uguaglianza non lineare inx.gatenta di soddisfareceq(x) = 0per tutte le voci di
ceq.
Per esempio,
x = ga(@myfun,4,A,b,Aeq,beq,lb,ub,@mycon)
dove mycon è una funzione MATLAB® come
function [c,ceq] = mycon(x) c = ... % Compute nonlinear inequalities at x. ceq = ... % Compute nonlinear equalities at x.
Per imparare a usare i vincoli vettoriali, vedere Vectorized Constraints .
Nota
ga non impone vincoli non lineari da soddisfare quando l'opzione PopulationType è impostata su 'bitString' o 'custom'.
Se intcon non è vuoto, il secondo output di nonlcon (ceq) deve essere una voce vuota ([]).
Per informazioni su come ga utilizza nonlcon, vedere Nonlinear Constraint Solver Algorithms for Genetic Algorithm.
Tipi di dati: char | function_handle | string
Opzioni di ottimizzazione, specificate come output di optimoptions o come struttura.
optimoptions nasconde le opzioni elencate in corsivo. Vedi Options that optimoptions Hides .
I valori in
{}indicano il valore predefinito.{}*rappresenta l'impostazione predefinita quando ci sono vincoli lineari e perMutationFcnanche quando ci sono limiti.I* indica l'impostazione predefinita per i vincoli interi oppure indica considerazioni speciali per i vincoli interi.
NM indica che l'opzione non si applica a
gamultiobj.
Opzioni per ga e gamultiobj
| Opzione | Descrizione | Valori |
|---|---|---|
ConstraintTolerance | Determina la fattibilità rispetto ai vincoli non lineari. Inoltre, Per una struttura di opzioni, utilizzare | Scalare non negativo | |
| Funzione che crea la popolazione iniziale. Specificare come nome di una funzione di creazione incorporata o di una funzione handle. Vedi Population Options . |
|
| Funzione utilizzata dall'algoritmo per creare children crossover. Specificare come nome una funzione crossover incorporata o una funzione handle. Vedi Crossover Options . |
|
| La frazione della popolazione nella generazione successiva, esclusi i children dell'élite, creata dalla funzione di crossover. | Scalare non negativo | |
| Livello di visualizzazione. |
|
| Funzione che calcola la misura della distanza degli individui. Specificare come nome una funzione di misura della distanza incorporata o una funzione handle. Il valore si applica alla variabile decisionale o allo spazio di progettazione (genotipo) o allo spazio funzionale (fenotipo). Il valore predefinito Per una struttura di opzioni, utilizzare una funzione handle, non un nome. |
|
| NM Numero intero positivo che specifica quanti individui della generazione attuale hanno la garanzia di sopravvivere alla generazione successiva. Non utilizzato in | Intero non negativo | |
| NM Se la funzione di fitness raggiunge il valore di | Scalare | |
| Funzione che scala i valori della funzione di fitness. Specificare come nome di una funzione di ridimensionamento integrata o di una funzione handle. Opzione non disponibile per |
|
FunctionTolerance | L'algoritmo si arresta se la variazione relativa media del valore della migliore funzione di fitness su Per Per una struttura di opzioni, utilizzare | Scalare non negativo | |
| I* Funzione che continua l'ottimizzazione dopo la terminazione di In alternativa, un array di celle che specifica la funzione ibrida e le sue opzioni. Vedi ga Hybrid Function . Per Quando il problema presenta vincoli interi, non è possibile utilizzare una funzione ibrida. | Nome funzione o handle | O Matrice di celle 1 per 2 | |
Penalità Iniziale | NM I* Valore iniziale del parametro di penalità | Scalare positivo | |
| Popolazione iniziale utilizzata per generare l'algoritmo genetico. Ha fino a Per una struttura di opzioni, utilizzare | Matrice | |
| Matrice o vettore che specifica l'intervallo degli individui nella popolazione iniziale. Si applica alla funzione di creazione Per una struttura di opzioni, utilizzare | Matrice o vettore | |
| Punteggi iniziali utilizzati per determinare l'idoneità. Ha fino a Per una struttura di opzioni, utilizzare | Vettore colonna per obiettivo singolo | matrice per multiobiettivo | |
| Numero massimo di iterazioni prima che l'algoritmo si arresti. Per una struttura di opzioni, utilizzare | Intero non negativo | |
| L'algoritmo si arresta se la variazione relativa media del valore della migliore funzione di fitness su Per Per una struttura di opzioni, utilizzare | Intero non negativo | |
| NM L'algoritmo si arresta se non si verifica alcun miglioramento nella funzione obiettivo per Per una struttura di opzioni, utilizzare | Scalare positivo |
| L'algoritmo si arresta dopo un'esecuzione di Per una struttura di opzioni, utilizzare | Scalare non negativo | |
Direzione della migrazione | Direzione della migrazione. Vedi Migration Options . |
|
Frazione di Migrazione | Scalare da 0 a 1 che specifica la frazione di individui in ciascuna sottopopolazione che migra verso una sottopopolazione diversa. Vedi Migration Options . | Scalare | |
Intervallo di migrazione | Numero intero positivo che specifica il numero di generazioni che hanno luogo tra le migrazioni di individui tra sottopopolazioni. Vedi Migration Options . | Numero intero positivo | |
| Funzione che produce mutation children. Specificare come nome una funzione di mutation incorporata o una funzione handle. Vedi Mutation Options . |
|
| Algoritmo di vincolo non lineare. Vedi Nonlinear Constraint Solver Algorithms for Genetic Algorithm . Opzione non modificabile per Per una struttura di opzioni, utilizzare |
|
| Funzioni che Per una struttura di opzioni, utilizzare | Funzione handle o array di celle di funzione handle | |
| Scalare da 0 a 1 che specifica la frazione di individui da mantenere sul primo fronte di Pareto mentre il risolutore seleziona gli individui dai fronti superiori, solo per | Scalare | |
Penalty Factor | NM I* Parametro di aggiornamento della penalità. | Scalare positivo | |
| Funzione che traccia i dati elaborati dall'algoritmo. Specificare come nome una funzione di grafico incorporata, una funzione handle o una matrice di celle di nomi incorporati o funzioni handle. Vedi Plot Options . Per una struttura di opzioni, utilizzare |
Solo Solo |
Intervallo di Trama | Numero intero positivo che specifica il numero di generazioni tra chiamate consecutive alle funzioni di tracciamento. | Numero intero positivo | |
| Dimensione della popolazione. | Intero positivo | |
| Tipo di dati della popolazione. Deve essere |
|
| Funzione che seleziona i genitori di crossover e mutation children. Specificare come nome di una funzione di selezione incorporata o di una funzione handle.
|
|
Test di Stallo | NM Interruzione del tipo di test. |
|
UseParallel | Calcolare in parallelo le funzioni di idoneità e di vincolo non lineare. Vedere Vectorize and Parallel Options (User Function Evaluation) e How to Use Parallel Processing in Global Optimization Toolbox . |
|
| Specifica se le funzioni sono vettorializzate. Vedere Vectorize and Parallel Options (User Function Evaluation) e Vectorize the Fitness Function . Per una struttura di opzioni, utilizzare |
|
Esempio optimoptions('ga','PlotFcn',@gaplotbestf)
Variabili intere, specificate come un vettore di numeri interi positivi che assumono valori da 1 a nvars. Ogni valore in intcon rappresenta un componente x che ha valore intero.
Nota
Se intcon non è vuoto, nonlcon deve restituire vuoto per ceq. Per ulteriori informazioni sulla programmazione intera, vedere Mixed Integer ga Optimization .
Esempio Per specificare che le voci pari in x hanno valore intero, impostare intcon su 2:2:nvars
Tipi di dati: double
Descrizione del problema, specificata come una struttura contenente questi campi.
fitnessfcn | Funzioni di fitness |
nvars | Numero di variabili di progettazione |
Aineq | Matrice |
Bineq | Vettore |
Aeq | Matrice |
Beq | Vettore |
lb | Limite inferiore su |
ub | Limite superiore su |
nonlcon | Funzioni di vincolo non lineare |
intcon | Indici di variabili intere |
rngstate | Campo per reimpostare lo stato del generatore di numeri casuali |
solver |
|
options | Opzioni create utilizzando |
È necessario specificare i campi fitnessfcn, nvars e options. I restanti sono facoltativi per ga .
Tipi di dati: struct
Argomenti di output
Soluzione, restituita come vettore reale. x è il punto migliore che ga ha individuato durante le sue iterazioni.
Valore della funzione obiettivo nella soluzione, restituito come numero reale. In genere, fval = fun(x) .
Motivo per cui ga si è fermato, è stato restituito come un numero intero.
| Bandiera di uscita | Significato |
|---|---|
1 | Senza vincoli non lineari — La variazione cumulativa media del valore della funzione di fitness su |
Con vincoli non lineari — L'entità della misura di complementarietà (vedere Misura di complementarietà) è minore di | |
3 | Il valore della funzione di fitness non è cambiato in |
4 | L'entità del passo è inferiore alla precisione della macchina e la violazione del vincolo è inferiore a |
5 | È stato raggiunto il limite minimo di idoneità |
0 | Superato il numero massimo di generazioni |
-1 | Ottimizzazione terminata da una funzione di output o da una funzione di grafico. |
-2 | Non è stato trovato alcun punto fattibile. |
-4 | Superato il limite di tempo di stallo |
-5 | Limite di tempo |
In presenza di vincoli interi, ga utilizza il valore di idoneità della penalità anziché il valore di idoneità per i criteri di arresto.
Informazioni sul processo di ottimizzazione, restituite come una struttura con questi campi:
problemtype— Tipo di problema, uno dei:'unconstrained''boundconstraints''linearconstraints''nonlinearconstr''integerconstraints'
rngstate— Stato del generatore di numeri casuali MATLAB, appena prima dell'avvio dell'algoritmo. È possibile utilizzare i valori inrngstateper riprodurre l'output diga. Vedi Reproduce Results .generations— Numero di generazioni calcolate.funccount— Numero di valutazioni della funzione di fitness.message— Motivo per cui l'algoritmo è terminato.maxconstraint— Violazione massima del vincolo, se presente.hybridflag— Flag di uscita dalla funzione ibrida. Si riferisce aHybridFcnoptions. Non applicabile agamultiobj.
Popolazione finale, restituita come matrice PopulationSize-by-nvars. Le righe di population sono gli individui.
Punteggi finali, restituiti come vettore colonna.
Per i problemi non interi, i punteggi finali sono i valori della funzione di idoneità delle righe di
population.Per i problemi interi, i punteggi finali sono i valori di idoneità di penalità dei membri della popolazione. Vedi Integer ga Algorithm .
Ulteriori informazioni
Nel risolutore di vincoli non lineari di Lagrangian aumentato, la misura di complementarietà è la norma del vettore i cui elementi sono ciλi, dove ci è la violazione del vincolo di disuguaglianza non lineare e λi è il moltiplicatore di Lagrange corrispondente. Vedi Augmented Lagrangian Genetic Algorithm .
Suggerimenti
Per scrivere una funzione con parametri aggiuntivi alle variabili indipendenti che possono essere chiamate da
ga, vedere Passing Extra Parameters.Per i problemi che utilizzano il tipo di popolazione
Double Vector(predefinito),ganon accetta funzioni i cui input sono di tipocomplex. Per risolvere problemi che coinvolgono dati complessi, scrivi le tue funzioni in modo che accettino vettori reali, separando la parte reale da quella immaginaria.
Algoritmi
Per una descrizione dell'algoritmo genetico, vedere Come funziona l'algoritmo genetico .
Per una descrizione dell'algoritmo di programmazione mista intera, vedere Integer ga Algorithm .
Per una descrizione degli algoritmi di vincolo non lineare, vedere Nonlinear Constraint Solver Algorithms for Genetic Algorithm .
Funzionalità alternativa
Applicazione
L'attività Ottimizza Live Editor fornisce un'interfaccia visiva per ga .
Riferimenti
[1] Goldberg, David E., Genetic Algorithms in Search, Optimization & Machine Learning, Addison-Wesley, 1989.
[2] A. R. Conn, N. I. M. Gould, and Ph. L. Toint. “A Globally Convergent Augmented Lagrangian Algorithm for Optimization with General Constraints and Simple Bounds”, SIAM Journal on Numerical Analysis, Volume 28, Number 2, pages 545–572, 1991.
[3] A. R. Conn, N. I. M. Gould, and Ph. L. Toint. “A Globally Convergent Augmented Lagrangian Barrier Algorithm for Optimization with General Inequality Constraints and Simple Bounds”, Mathematics of Computation, Volume 66, Number 217, pages 261–288, 1997.
Funzionalità estese
Per l'esecuzione in parallelo, impostare l'opzione 'UseParallel' su true.
options = optimoptions('solvername','UseParallel',true)
Per ulteriori informazioni, vedere How to Use Parallel Processing in Global Optimization Toolbox .
Cronologia versioni
Introduzione prima di R2006aQuando la funzione di fitness è deterministica, ga non rivaluta la funzione di fitness sugli individui d'élite (i migliori al momento). È possibile controllare questo comportamento accedendo al nuovo campo state.EvalElites e modificandolo in una funzione di output personalizzata o in una funzione di grafico personalizzata. Allo stesso modo, quando la popolazione iniziale presenta membri duplicati, ga valuta ogni membro univoco solo una volta. È possibile controllare questo comportamento in una funzione di output personalizzata o in una funzione di grafico personalizzata accedendo e modificando il nuovo campo state.HaveDuplicates. Per maggiori dettagli, vedere Custom Output Function for Genetic Algorithm o Funzione grafico personalizzata.
Per maggiori dettagli sui due nuovi campi, vedere The State Structure .
MATLAB Command
You clicked a link that corresponds to this MATLAB command:
Run the command by entering it in the MATLAB Command Window. Web browsers do not support MATLAB commands.
Seleziona un sito web
Seleziona un sito web per visualizzare contenuto tradotto dove disponibile e vedere eventi e offerte locali. In base alla tua area geografica, ti consigliamo di selezionare: .
Puoi anche selezionare un sito web dal seguente elenco:
Come ottenere le migliori prestazioni del sito
Per ottenere le migliori prestazioni del sito, seleziona il sito cinese (in cinese o in inglese). I siti MathWorks per gli altri paesi non sono ottimizzati per essere visitati dalla tua area geografica.
Americhe
- América Latina (Español)
- Canada (English)
- United States (English)
Europa
- Belgium (English)
- Denmark (English)
- Deutschland (Deutsch)
- España (Español)
- Finland (English)
- France (Français)
- Ireland (English)
- Italia (Italiano)
- Luxembourg (English)
- Netherlands (English)
- Norway (English)
- Österreich (Deutsch)
- Portugal (English)
- Sweden (English)
- Switzerland
- United Kingdom (English)